3月3日,《基因组蛋白质组与生物信息学报》公布了2022年度“中国生物信息学十大进展”评选结果,西安交通大学自动化学院叶凯教授团队成果“基因组复杂结构变异检测方法——SVision”入选。
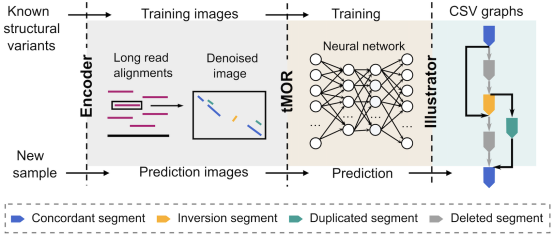
基因组结构变异是物种进化和疾病发生的重要驱动因素,对结构变异的全面精准检测表征是基因组精细结构研究的核心环节,然而目前尚缺乏针对基因组重复区域复杂结构变异的检测策略。叶凯教授团队针对基因组重复区域背景噪声高、复杂结构变异类型未知且建模难,通过将基因组结构变异检测从序列空间转换为图像空间,实现了简单和复杂类型结构变异的高性能检测和准确表征。团队开发了基于深度学习的多目标识别方法SVision,该方法无需依赖先验信息,能够从长读长测序数据中自动检测和表征未知类型的基因组结构变异,为后续多种生物医学应用场景的全类型基因组结构变异研究提供了有力工具和新方案。该成果发表于《自然-方法》(Nature Methods)杂志。
原文链接:https://www.nature.com/articles/s41592-022-01609-w
工具链接:https://github.com/xjtu-omics/SVision
《基因组蛋白质组与生物信息学报》(简称GPB)于2003年创刊,是由中国科学院主管、中国科学院北京基因组研究所(国家生物信息中心)与中国遗传学会共同主办的英文学术期刊,由Elsevier金色开放获取(Gold Open Access)出版。刊载来自世界范围内组学、生物信息学及相关领域的优质稿件。现为中国科学引文数据库(CSCD)和中国科技论文与引文数据库(CSTPCD)核心期刊,被SCIE、PubMed / MEDLINE、Scopus等数据库收录。









